지난 글에서는 초기 코로나 바이러스의 DNA 패턴을 분석하는 방법으로 k-mers 라는 부분 문자열로 나누어서 분석하는 방법을 알아보았다. 지난 글 마지막에서 보았을 때는, 초기 코로나 바이러스의 스파이크 단백질 염기 서열을 알고 있었고, 그 단백질의 염기서열을 코돈으로 번역하여 아미노산 서열을 찾아보았다.
하지만 실제로는 지난 글과 같이 '염기서열을 알고있는' 상태에서 찾는 것이 아니라, 염기 서열 안에서 유전자를 찾아야 하는 상황이 더 많다.
만약 우리가 찾아야 하는 유전자의 염기 서열을 모를 때 유전자를 어떻게 찾을 수 있을까?
우리는 모든 유전자가 특정 코돈 (AUG) 으로 시작하고 특정 코돈들 중 하나 (TAA, TAG, TGA) 로 끝나는 것을 알고있다.
그렇다면 개시 코돈과 종결 코돈만으로 유전자를 찾을 수 있을까?
아쉽게도 이것만으론 쉽지가 않다.
먼저 개시코돈이 매우 자주 등장하므로 우리가 찾으려는 유전자가 어떤 개시코돈에서 시작해야하는지 알 수 없고,
무엇보다 진핵생물의 경우, DNA 슬라이싱이 빈번하게 일어나서, 뒤에있는 개시코돈에서 시작한 서열이 그 이전의 염기서열과 결합한 뒤 종결코돈으로 끝나는 현상도 발생한다.

따라서 단순히 개시코돈 3개 만으로는 유전자를 알 수 없고, 다양한 조합을 시도해봐야 한다.
그렇다면 실제로 우리 몸에서는 DNA 염기서열로부터 어떻게 유전자를 찾아낼까?
유전자 전사에 관여하는 RNA 중합 효소는 유전자 프로그래밍 영역의 상류에 해당하는 regulatory region 이라는 곳에서 '전사 인자' 라고 부르는 단백질과 함께 전사에 관여한다. 이 단백질은 regulatory region 을 찾고, RNA 중합효소를 끌어당긴다.
regulatory region 에는 TFBS 라는 것이 존재하며, motif 라고 부르는, 전사 인자에 의해 특정된 특별한 DNA 서열이 존재한다.
하나의 전사인자는 여러개의 유전자 발현에 관여할 수 있다.
또한 사람 유전자의 10% 정도가 전사인자에 대한 유전자이다.
하지만 우리는 모든 전사인자가 특정하는 각각의 motif 를 알지 못하고, TSBF 가 어디있는지도 모른다.
또한 유전자마다 motifs 도 조금씩 다를 수 있으며, 그저 motif 가 어딘가에 있다는 것만 알고 있다.

다음 10개의 DNA 시퀀스에서 공통적으로 한번씩만 나타나는 10-mer 을 찾아보자.

탐색 결과는 위와 같다.
과연 어떻게 찾을 수 있을까?
1. 브루트 포스
하나의 문자열에서 모든 10-mer 을 시도하고,
각각의 시도마다 다음 문자열에서 모든 10-mer 을 시도하고,
또 그 각각의 시도마다 다음 문자열에서 모든 10-mer 을 시도하고,...
를 M 개의 문자열에 대해 반복한다.
하나의 문자열의 길이가 N일 때 이 문자열에는 N - k + 1 개의 k-mer 들이 있다.
모든 k-mer 에 대해서 M 개의 문자열에 대해 모든 조합을 시도해보는 경우의 수는
(N - k + 1) ** M 이다.

코드로는 위와 같이 구현할 수 있다.
dna 에는 M 개의 DNA 문자열들이 주어지며, 각각의 DNA 문자열 길이는 N 이다.
파이썬의 itertools 가 제공하는 product 메서드를 사용하여 0부터 N-k+1 미만의 범위의 수를 M 번 반복적으로 선택한다. (중복순열)
이를 offset 으로 삼아서 각 dna 문자열에 대해 이 오프셋을 기준으로 k-mer을 찾은 뒤, 만약 10개의 k-mer 가 모두 동일하다면 그 offset 을 반환하도록 만든다.

4개의 DNA 문자열에 대해 공통된 10-mer 을 찾는 프로그램을 돌렸더니 6.25초 뒤에 답을 발견했다.
Consensus

이번엔 조금 더 현실적인 문제를 살펴보자.
10개의 10-mer 가 정확하게 일치할 필요는 없고, '유사하기만' 하면 된다.
이 각각의 k-mers에는 우리가 찾으려는 motif 와 정확하게 일치하는 k-mer가 없을 수도 있다.
어떻게 하면 이렇게 유사한 motifs 들을 찾을 수 있을까?
직관적인 방법은, 모든 motif 쌍 조합을 다 시도해보고 얼마나 매칭되는지 계산한 뒤, 그 중 많이 매칭되는 조합을 고르는 것이다.
또 다른 방법은 각 문자열에서 k-mer를 하나씩 뽑아서 조합을 만들고, 그 조합에 대해 consensus 점수를 매겨서 제일 높은 점수를 갖는 consensus를 찾는 것이다.

방법은 간단하다. 위 그림처럼 5개의 8-mer 들이 있다고 하면, 이들을 모두 나란히 정렬한 뒤, 각 위치마다 ACGT의 개수를 센다.
그리고 제일 많이 등장한 염기를 Consensus 의 염기로 만들어서 구하는 것이다.
이때 각 Alignment 를 구성하는 경우의 수를 해볼 때는 각 DNA 서열에서 모든 k-mer 조합을 다 시도해봤던 그 방법으로 구해서 Consensus를 찾는다.
이렇게 구한 Consensus motif 를 'ancestor motif' 라고 부른다.
Consensus motif 와 실제 motif 사이의 '거리' 는 일반적으로 그 어떤 실제 motif 2개의 거리보다도 가깝다.
이때 두 문자열 사이의 '거리' 를 잴 때는 Hamming Distance 를 사용한다.
Hamming Distance 는 두 문자열 사이의 서로 다른 문자의 개수를 말한다.

이때 consensus string 은 이 문자를 구하는데 사용된 모든 k-mer 사이의 평균 Hamming Distance가 최소이다.

ACGTACGT 라는 Consensus string 은 이를 구하는데 사용된 CCGTACGG, AGGTACTT ... 와 같은 문자열들 사이의 hamming distance 를 구했을 대 그 평균이 최소가 된다.
이제 이 과정을 코드로 구현해보자.

어떤 DNA 서열 하나에 대한 점수는 위와 같이 계산한다.
DNA 서열의 위치 1 .. k 에 대해 ACGT 의 개수를 세서 제일 개수가 많은 것을 취하고, 그 개수들의 합이 점수가 된다.
그리고 가능한 DNA 서열들에 대해 이를 반복하면서 점수를 계산한다.
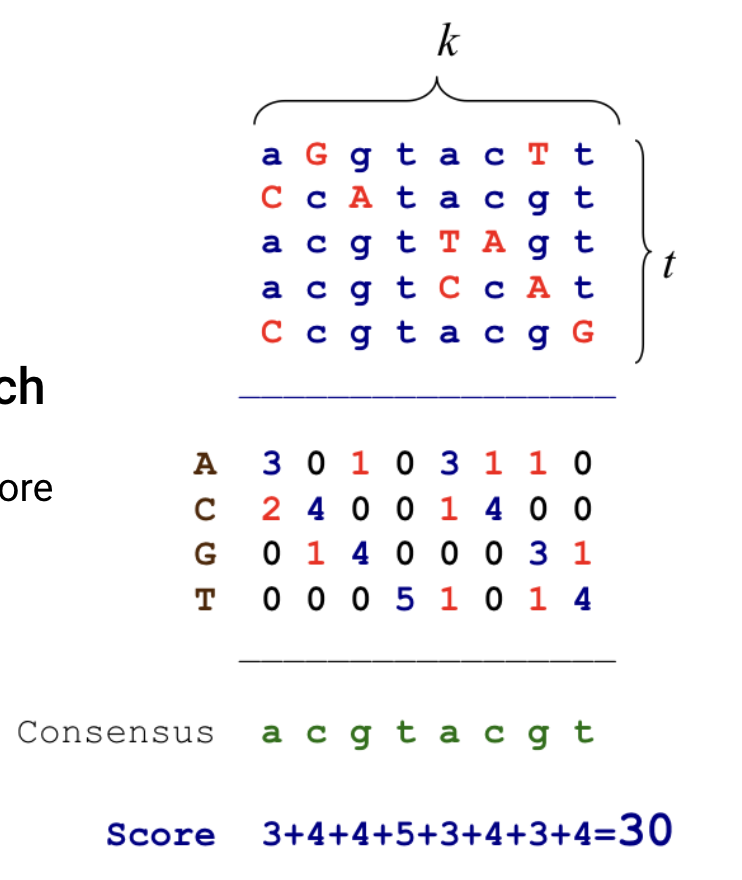
위에서 구한 consensus 에 대해 점수를 매겨보면 위와 같이 30점의 점수가 나온다.
이제 기존의 Bruteforce 방법을 통한 motif 탐색 방법을 정확히 일치하는 motif 가 아니라 유사한 motif 를 찾을 수 있도록 수정해보자.

특정 DNA 서열에 대해 점수를 계산하는 함수를 위와 같이 작성한다.
S 에는 M 개의 DNA 부분문자열에 대한 각각의 offset(시작 위치)이 튜플로 주어진다.

기존의 BruteForceMotifSearch 함수를 위와 같이 점수를 계산해서 점수가 제일 높은 motif 를 얻도록 한다.
그리고 가장 높은 점수를 얻은 offset 과 그때의 점수를 반환한다.

위와 같은 데이터가 주어졌을 때 13분 정도의 시간이 지난 뒤 36점이라는 최고 점수와 그 점수가 나왔던 offset 들을 알 수 있다.
시간 복잡도
그런데 브루트포스 알고리즘을 이용한 motif search 는 시간이 너무 오래 걸린다.
우선 M개의 DNA 문자열에 대해 각각의 모든 k-mer 조합을 다 해보는 것은 (N - k + 1) ** M 의 시간이 걸린다.
(N : 각 DNA 문자열의 길이)
그리고 각각에 대해 점수를 계산해야하는데, Score 함수는 Mk 시간복잡도가 걸린다.
길이가 k 인 부분 문자열 M 개를 모두 순회해야 하기 때문이다.
따라서 전체 시간 복잡도는

이렇게 된다.
만약 M = 10, N = 80, k = 10 이라면, 대략 10**21 의 계산을 해야 한다.
컴퓨터가 1초에 10**9 번 연산을 할 수 있다고 가정하면, 10**12 초 동안 연산을 해야하며, 이를 연으로 환산하면 무려 3만 년이 넘는다.
게다가 위에서는 1초에 10억번 연산을 '가정' 했지만, 실제로 파이썬을 돌려보면 초당 585만번 정도의 연산을 수행한다.
따라서 실제로는 위에서 예측한 시간보다 훨씬 오래 걸린다.
그 이유 중 하나는 itertools 패키지에서 제공하는 product 함수의 실행 시간이 지수적으로 증가하기 때문이다.
그래서 이 함수는 불필요한 코드 작성을 줄여주는 도구인 동시에, 계산 시간을 지연시키는 장애물이 되기도 한다.
우리가 찾으려는 motif 를 알고 있을 때는 M 개의 문자열에 대해서 N - k + 1 개의 k-mer 을 우리가 알고있는 motif 와 같은지 검사하면 되고, 각각의 검사에는 k 번의 문자열 비교가 필요하므로

의 시간복잡도가 걸린다.
다음 글에서는 어떻게하면 조금 더 빠르게 원하는 motif 를 찾을 수 있는지 고민해본다.
'CS > 문제해결기법' 카테고리의 다른 글
| 6. Advanced Sequence Alignment (0) | 2024.10.29 |
|---|---|
| 5. Aligning Sequences (0) | 2024.10.29 |
| 4. Finding TFBS Motifs in our lifetime (0) | 2024.10.29 |
| 2. Genome (2) | 2024.10.28 |
| 1. Bioinformatics (1) | 2024.10.28 |
